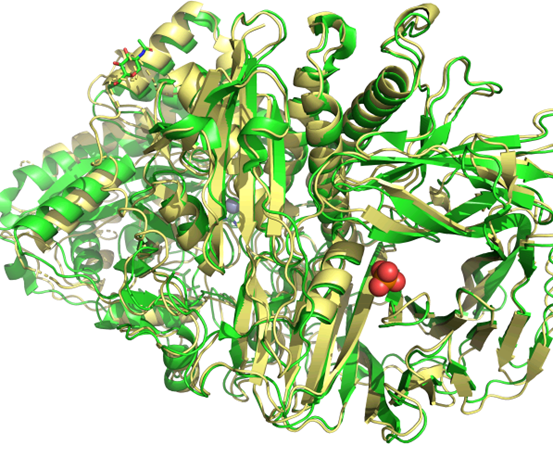
一·、在北极星集群上提交RoseTTAFold-All-Atom------PDB格式
库存放在/lustre3/share/RoseTTAFold-All-Atom
RFAA环境:source /appsnew/source/RFAA.sh
pymol环境:source /appsnew/source/pymol3.sh
## pymol和RFAA 冲突不能同时安装
RFAA.run 集成了上述得所有环境,无需source ,用原生得就用上述环境
一、命令格式
RFAA.run [–pymol] [pdb|fasta] [small.pdb|small.mol2]
格式说明
1、 直接跑 pdb结构 : RFAA.run 1tce.pdb
如果想只跑A链 RFAA.run 1tce.pdb:A
2、将所有的蛋白、DNA、RNA合成一个fasta文件 : RFAA.run input.fasta
!!!!这里多个蛋白或核酸链,fasta文件必须合一个文件,会被自动切割几个文件
3、 跑小分子mol2格式 : RFAA.run 1tce.pdb XG4.mol2
RFAA.run input.fasta XG4.mol2
4、 跑小分子pdb格式 : RFAA.run 1tce.pdb XG4.pdb
RFAA.run input.fasta XG4.pdb
5、 跑小分子sdf格式(OE生成的): RFAA.run 1tce.pdb XG4.sdf
openeye(含smiles格式,推荐4,5) RFAA.run input.fasta XG4.sdf
6、 生成pymol的pse文件,和): RFAA.run -pymol1tce 1tce.pdb XG4.sdf
RFAA.run -pymol 1tce 1tce.pdb
7、共价,预生成格式: RFAA.run 1tce.pdb XG4.pdbyaml
共价键的yaml文件生成请参考 https://github.com/baker-laboratory/RoseTTAFold-All-Atom
修改后提交同上
-h, --help 显示帮助信息
-v, --version 显示版本信息
二、提交案例-pdb
案例路径:/gpfs3/database/RFAA/alian/2JO9
1、预测已经有的PDB结构
1)提交到gpu_4l(800氨基酸以下):pkurun-g4c 1 1 RFAA.run -pymol2JO9 2JO9.pdb:A
其中-pymol2JO9,表示跑完后会和2JO9结构alian并生成pse文件和log文件
生成的脚本如下:
(pymol3) [chen@login28 testRFAA-v100]$ cat job.srp234634
#!/bin/bash
#SBATCH -J RFA234634
#SBATCH -p gpu_4l
#SBATCH -N 1
#SBATCH -o RFA234634_%j.out
#SBATCH -e RFA234634_%j.err
#SBATCH --no-requeue
#SBATCH -A chen_g1
#SBATCH --qos=cheng4l
#SBATCH --gres=gpu:1
#SBATCH --overcommit
#SBATCH --mincpus=7
RFAA.run -pymol2JO9 2JO9.pdb:A
如果想只跑A链: pkurun-g4c 1 1 RFAA.run 2JO9.pdb
2)提交到gpu_2l (800氨基酸以下):pkurun-g2c 1 1 RFAA.run -pymol2JO9 2JO9.pdb:A
3)提交到gpu_a800(1300氨基酸以下):pkurun-a800 1 1 RFAA.run -pymol2JO9 2JO9.pdb:A
3)提交到gpu_ah00(1300氨基酸以下):pkurun-h800 1 1 RFAA.run -pymol2JO9 2JO9.pdb:A
三、结果
1、生成的文件
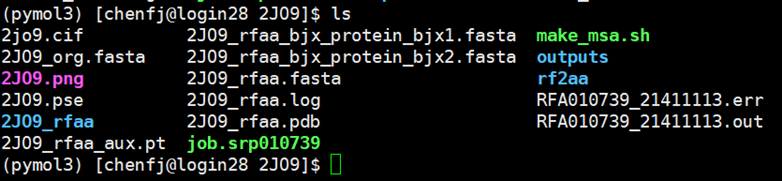
2JO9.png为绘图文件,2JO9_rfaa.log 为日志文件,2JO9_rfaa_aux.pt为torch文件,2JO9_org.fasta为pdb原始fasta文件,2JO9.pse为pymol文件,可以用pymol直接打开
yaml文件:
(pymol3) [chen@login28 2JO9]cat rf2aa/config/inference/2JO9_rfaa.yaml
#gen yaml
###bjx ./2JO9_rfaa_bjx_protein_bjx1.fasta
defaults:
- base##基本的定义,路径
job_name: "2JO9_rfaa"
protein_inputs:
A:
fasta_file: ./2JO9_rfaa_bjx_protein_bjx1.fasta
2、2JO9_rfaa.log文件
(pymol3) [chen@login28 2JO9]$ cat 2JO9_rfaa.log
pae_inter: nan ##为相互作用的打分13分以下为佳
mean_plddt: 0.7583932876586914
mean_pae: 12.079401016235352
pae_prot: 12.079401016235352
#gen pymol by chenfj
2JO9: (1.526375412940979, 29, 3, 1.8148088455200195, 31, 213.0, 37) ##红色为RMSD
runned min 5.62m ## 跑的时间
3、打开2JO9.pse文件(2JO9.png为绘图)
使用北极星图形界面(web)直接打开来看,如下
四:不同长度蛋白质序列在不同GPU上的测试:
PDB | 2JO9 | 3FDT | 3DGL | 1TCE | 3B2U | 3POV | 3AY6 | 3DB6 | 1OFG | 1MUU | 1E7M | 2V5P | 1CF3 |
aa number | 33 | 60 | 82 | 108 | 225 | 229 | 270 | 298 | 382 | 437 | 486 | 493 | 584 |
V100(min) | 3.68 | 108.33 | 107,51 | 108.33 | 23.51 | 115.02 | 17.35 | 25.18 | 12.47 | 11.38 | 8.02 | 90.47 | 15.27 |
A800(min) | 2.78 | 112.17 | 106.05 | 108.05 | 22.45 | 118.3 | 18.57 | 29.68 | 11.73 | 11.63 | 5.28 | 13.48 | 13.29 |
H800(min) | 7.35 | 73.85 | 72.93 | 72.38 | 22.05 | 79.15 | 21.35 | 30.43 | 10.82 | 10.28 | 4.75 | 47.82 | 12.25 |
RMSD(埃) | |||||||||||||
V100 | 1.569 | 0.611 | 0.727 | 0.889 | 0.983 | 1.925 | 0.402 | 0.692 | 0.491 | 4.815 | 1.394 | 1.215 | 1.219 |
A800 | 0.8 | 0.468 | 0.684 | 0.934 | 1.294 | 2.161 | 0.383 | 0.7 | 0.522 | 6.259 | 1.142 | 1.374 | 0.505 |
H800 | 1.566 | 0.463 | 0.679 | 0.874 | 1.063 | 1.634 | 0.37 | 0.752 | 0.514 | 20.526 | 1.298 | 12.76 | 0.528 |
PDB | 6HN1 | 4A5S | 4ANU | 6EQC | 7LZH | 5L6H | 3D4Y | 2F18 | 1G9A | 4XWK | 1T3T | 5KIS | 5URK | 6MU1 |
aa number | 610 | 741 | 833 | 953 | 960 | 1029 | 1046 | 1046 | 1291 | 1295 | 1304 | 1492 | 1739 | 2740 |
V100(min) | 73.15 | 30.9 | 显存超了V100 | |||||||||||
A800(min) | 103.46 | 29.98 | 106.28 | 102.1 | 15.03 | 108.29 | 9.6 | 11.12 | 93.36 | 49.51 | 显存超了H800 | |||
H800(min) | 29.82 | 29.87 | 46.15 | 46.38 | 14.67 | 54.5 | 9 | 9.03 | 50.3 | 50.43 | 显存超了H800 | |||
RMSD(埃) | ||||||||||||||
V100 | 3.197 | 17.972 | 显存超了V100 | |||||||||||
A800 | 1.321 | 8.049 | 5.654 | 5.595 | 41.84 | 1.284 | 2.065 | 26.87 | 6.189 | 31.92 | 显存超了H800 | |||
H800 | 1.511 | 7.697 | 7.609 | 12.801 | 39.421 | 1.057 | 6.225 | 1.823 | 14.407 | 29.674 | 显存超了H800 | |||
表上结果显示A800和H800并没有比V100快多少,而且V100最好不要超过800氨基酸,A800和H800不要超过1300,当氨基酸残基超过650个的时候RFAA的RMSD会变得异常,远不如AF2.3,不过速度会快。
结构示例:
2JO9
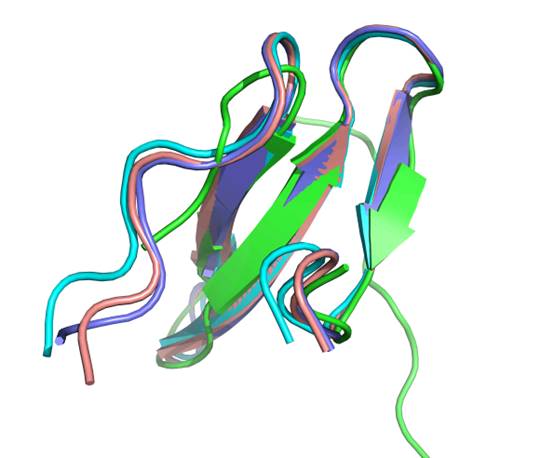
4A5S-a800
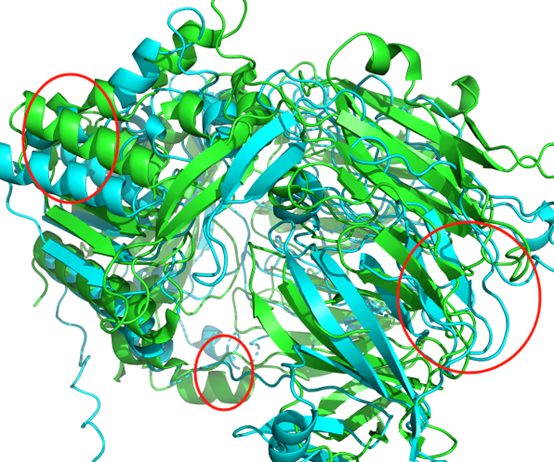
4A5S-V100
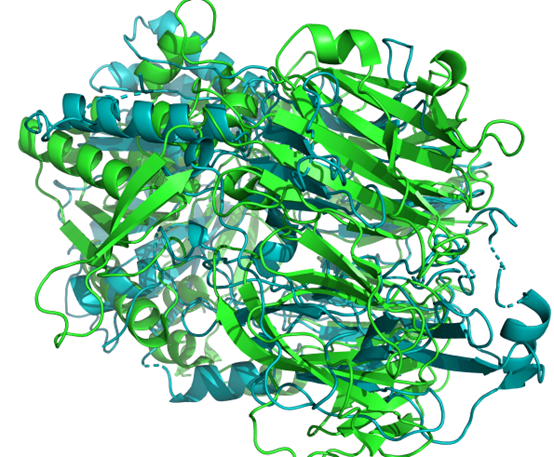
4ANU
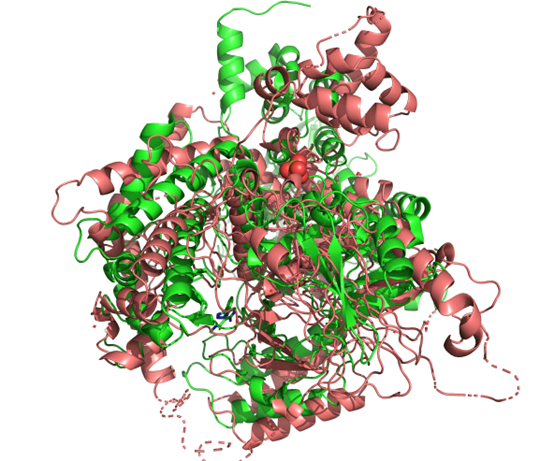
2F18-a800—rmsd 26.872
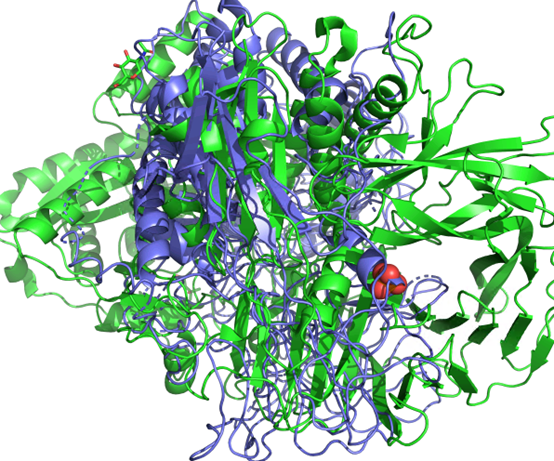
2F18-h800—rmsd 1.823