三、在北极星集群上提交RoseTTAFold-All-Atom------核酸序列
一、fasta格式,必须按照标准来定义:
1、核酸和蛋白的fasta格式:
8IK4_org.fasta:
>8IK4_1|Chains A, B|Type IV methyl-directed restriction enzyme EcoKMcrB subunit|Escherichia coli K-12 (83333)
MESIQPWIEKFIKQAQQQRSQSTKDYPTSYRNLRVKLSFGYGNFTSIPWFAFLGEGQEASNGIYPVIFYYKDFDELVLAYGISDTNEPHAQWQFSSDIPKTIAEYFQATSGVYPKKYGQSYYACSQKVSQGIDYTRFASMLDNIINDYKLIFNSGKSVIPPLEGHHHHHH
>8IK4_2|Chain C|DNA (5'-D(*AP*GP*AP*(5CM)P*CP*GP*GP*TP*AP*G)-3')|DNA molecule (2853804)
TGAGACCGGTAGC
>8IK4_3|Chain D|DNA (5'-D(*CP*TP*AP*CP*CP*GP*GP*TP*CP*T)-3')|DNA molecule (2853804)
AGCTACCGGTCTC
二、命令格式
RFAA.run [–pymol] [pdb|fasta] [small.pdb|small.mol2]
格式说明
1、 直接跑 pdb结构 : RFAA.run 1tce.pdb
如果想只跑A链 RFAA.run 1tce.pdb:A
2、将所有的蛋白、DNA、RNA合成一个fasta文件 : RFAA.run input.fasta
!!!!这里多个蛋白或核酸链,fasta文件必须合一个文件,会被自动切割几个文件
3、 跑小分子mol2格式 : RFAA.run 1tce.pdb XG4.mol2
RFAA.run input.fasta XG4.mol2
4、 跑小分子pdb格式 : RFAA.run 1tce.pdb XG4.pdb
RFAA.run input.fasta XG4.pdb
5、 跑小分子sdf格式(OE生成的): RFAA.run 1tce.pdb XG4.sdf
openeye(含smiles格式,推荐4,5) RFAA.run input.fasta XG4.sdf
6、 生成pymol的pse文件,和): RFAA.run -pymol1tce 1tce.pdb XG4.sdf
RFAA.run -pymol 1tce 1tce.pdb
7、共价,预生成格式: RFAA.run 1tce.pdb XG4.pdbyaml
共价键的yaml文件生成请参考 https://github.com/baker-laboratory/RoseTTAFold-All-Atom
修改后提交同上
-h, --help 显示帮助信息
-v, --version 显示版本信息
三、提交案例-fasta
案例路径:/gpfs3/database/RFAA/alian
1、建立并保存fasta: 8IK4_rfaa.fasta
>8IK4_1|Chains A, B|Type IV methyl-directed restriction enzyme EcoKMcrB subunit|Escherichia coli K-12 (83333)
MESIQPWIEKFIKQAQQQRSQSTKDYPTSYRNLRVKLSFGYGNFTSIPWFAFLGEGQEASNGIYPVIFYYKDFDELVLAYGISDTNEPHAQWQFSSDIPKTIAEYFQATSGVYPKKYGQSYYACSQKVSQGIDYTRFASMLDNIINDYKLIFNSGKSVIPPLEGHHHHHH
>8IK4_2|Chain C|DNA (5'-D(*AP*GP*AP*(5CM)P*CP*GP*GP*TP*AP*G)-3')|DNA molecule (2853804)
TGAGACCGGTAGC
>8IK4_3|Chain D|DNA (5'-D(*CP*TP*AP*CP*CP*GP*GP*TP*CP*T)-3')|DNA molecule (2853804)
AGCTACCGGTCTC
2、yaml文件
cat rf2aa/config/inference/8IK4_rfaa.yaml
#gen yaml
###bjx ./8IK4_rfaa_bjx_protein_bjx1.fasta
###bjx ./8IK4_rfaa_bjx_dna_bjx2.fasta
###bjx ./8IK4_rfaa_bjx_dna_bjx3.fasta
defaults:
- base
job_name: "8IK4_rfaa"
protein_inputs:
A:
fasta_file: ./8IK4_rfaa_bjx_protein_bjx1.fasta
na_inputs:
B:
fasta: ./8IK4_rfaa_bjx_dna_bjx2.fasta
input_type: "dna"
C:
fasta: ./8IK4_rfaa_bjx_dna_bjx3.fasta
input_type: "dna"
3、提交任务
1)提交到gpu_4l (800氨基酸以下): pkurun-g4c 1 1 RFAA.run -pymol 8IK4 8IK4_rfaa.fasta
提交脚本
#!/bin/bash
#SBATCH -J RFA002851
#SBATCH -p gpu_4l
#SBATCH -N 1
#SBATCH -o RFA002851_%j.out
#SBATCH -e RFA002851_%j.err
#SBATCH --no-requeue
#SBATCH -A chen_g1
#SBATCH --qos=cheng4c
#SBATCH --gres=gpu:1
#SBATCH --overcommit
#SBATCH --mincpus=7
pkurun RFAA.run -pymol 8qhh 8qhh_input.fasta
2)提交到gpu_2l (800氨基酸以下): pkurun-g2c 1 1 RFAA.run -pymol 8IK4 8IK4_rfaa.fasta
3)提交到gpu_a800(1300氨基酸以下):pkurun-a800 1 1 RFAA.run -pymol 8IK4 8IK4_rfaa.fasta
3)提交到gpu_ah00(1300氨基酸以下):pkurun-h800 1 1 RFAA.run -pymol 8IK4 8IK4_rfaa.fasta
四、结果
1、对log文件分析
(pymol3) [chen@login28 alian]$ cat 8IK4_rfaa.log
#gen pymol by chenfj
8IK4: (0.39987727999687195, 143, 5, 2.8700876235961914, 159, 922.0, 170) ##红色为RMSD
pae_inter: nan
mean_plddt: 0.8007603883743286
mean_pae: 9.388115882873535
pae_prot: 9.388115882873535)
runned min 44.5m ## 跑的时间
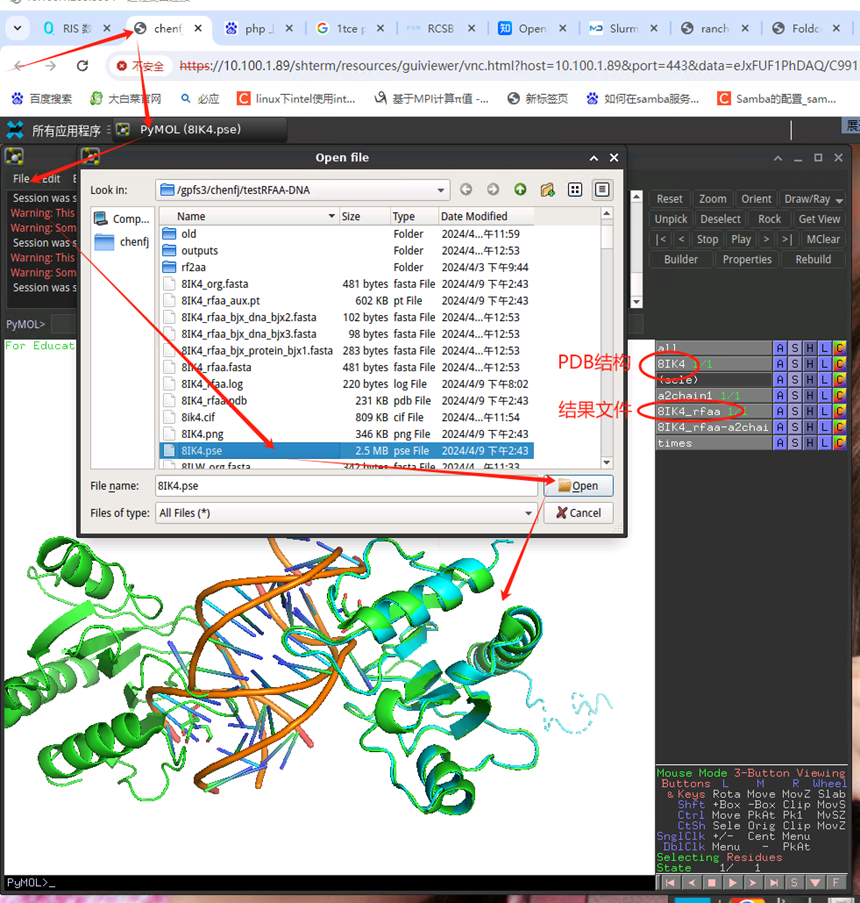
2、打开pse文件
使用北极星图形界面(web)直接打开来看,如下
3、其他结构
PDB | 6I1V:6I1W | 6IJ2 | 6JVX | 6KR6 | 6KTC | 6L0Y | 6L5N |
分类 | ssRNA | RNA | RNA | RNA | RNA | RNA | RNA |
报错 | Class values must be smaller than num_classes | CG拆分了 | Class values must be smaller than num_classes | need at least one array to concatenate | Class values must be smaller than num_classes | Class values must be smaller than num_classes | Class values must be smaller than num_classes |
特征序列 | UCACUGAGU | CG | UGUGUGUGUGUG | UAUUN | GCCU | GGUAGACGCUUCGGCGUUUGCC | UUUUUUUUUUUUUUU |
PDB | 8IK4 | 8ILW | 8JFR | 8JFU | 8JIC | 8JKK | 8K8D |
分类 | DNA、PRO | DNA、PRO | DNA、PRO | DNA、PRO | DNA | DNA、PRO | DNA、PRO |
报错 | 可 | DNA偏移 | DNA散了 | 都散了 | 乱了 | 散了 | 分了 |
特征序列 | |||||||
RMSD(pro) | 0.37 | 0.424 | 0.49 | 15.683 | 15.945 | 0.626 | 0.852 |
核酸结构 | TGAGACCGGTAGC | CAACACTTAATCCAAA | ATTATGACAAATGTCATAG | ATTATGACAAATGTCATAG | TAGGGTTAGGGTTAGGGTTAGG | CGATCCGCTACG | CATTACGTAATG |
时间(分钟) | 19.37 | 4.05 | 7.18 | 37.27 | 0.25 | 31.4 | 5.53 |
PDB | 8RZV | 8S1W | 8U0O | 8U8L | 8VXA | 8XA9 |
分类 | DNA、PRO | DNA、PRO | DNA、PRO | DNA、PRO | DNA | DNA、PRO |
报错 | 散了 | 散了 | 散了 | 可 | 散了 | 偏了一条链 |
特征序列 | CGACCGCCAGC | |||||
RMSD(pro) | 0.89 | 18.676 | 1.099 | 0.625 | 4.44 | 0.625 |
核酸结构 | TTAGGGTTAGGG | GGGCGCGAAGCATTCGCGGGGTTAGGGTTAGGG | ACGCGGCA | CTGTTAGGCTTAGGCTTAG | TCGTCACCAGTACAAACTACAACGCCTGTAGCATTCCACA | TTTTTTTGCTGGCGGTCG |
时间(分钟) | 8.18 | 0.25 | 5.07 | 88.73 | 93.35 | 63.33 |
核酸跑的很差,RNA和DNA单链目前还没有涉及,DNA双链也就是很少一部分能跑的可以
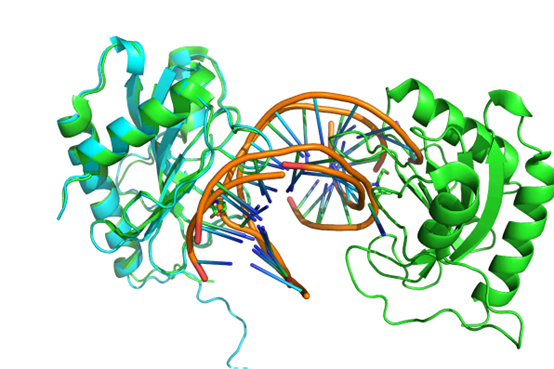
五、结果展示:
8IK4
6IJ2
8ILW
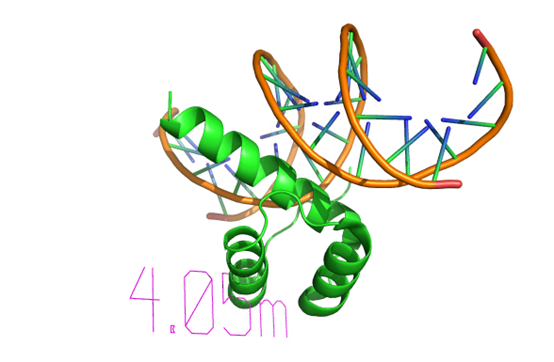
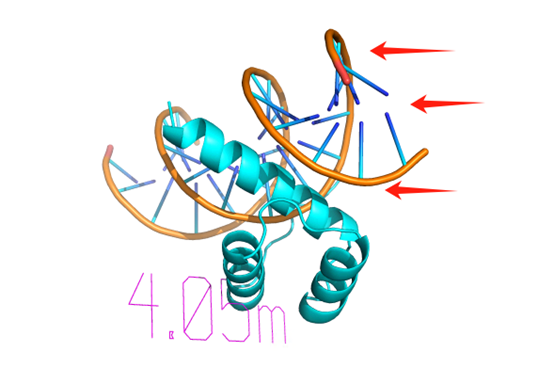
8JFR
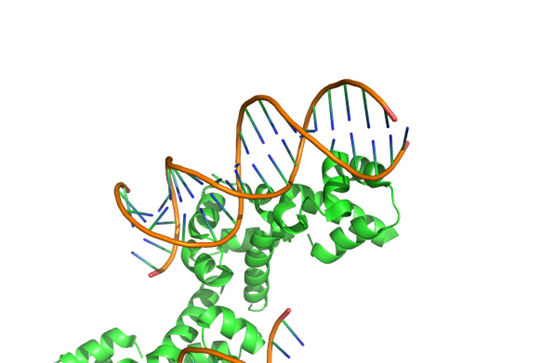
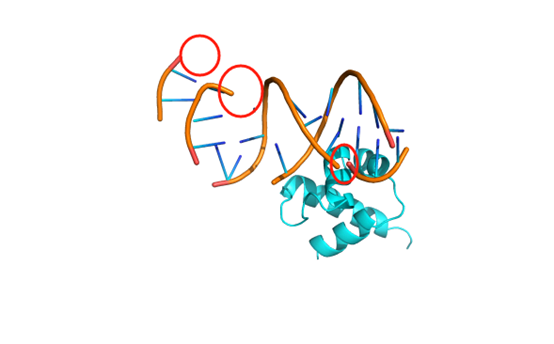
8JFU
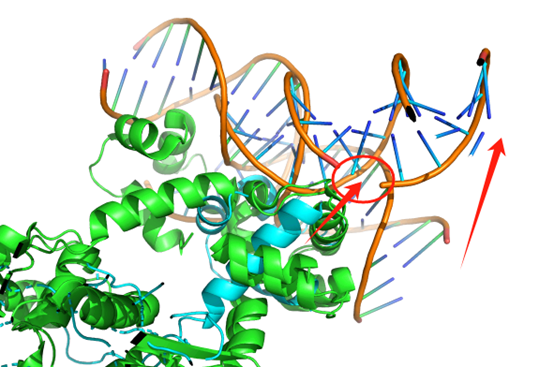
8JIC
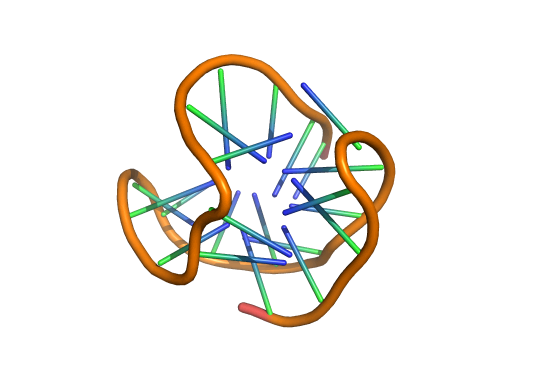
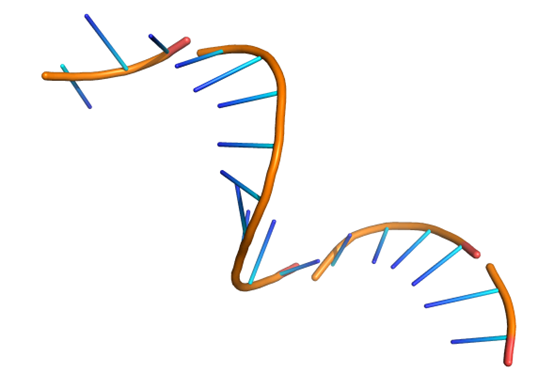
8JKK
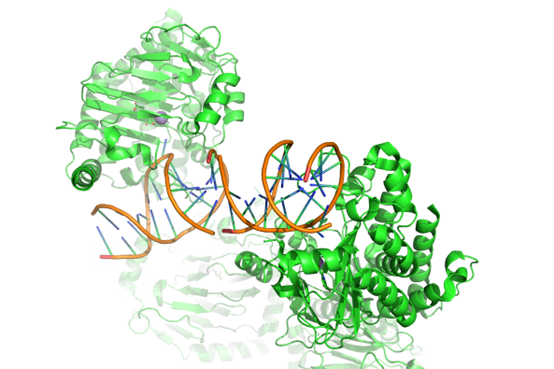
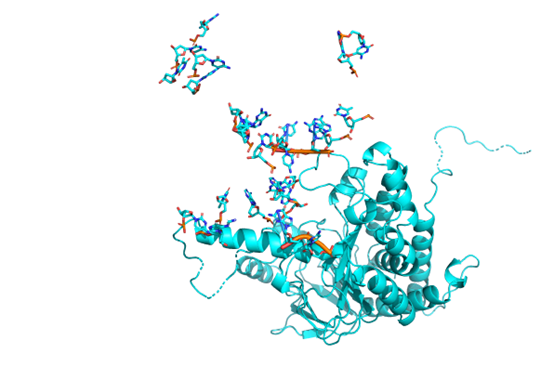
8K8D
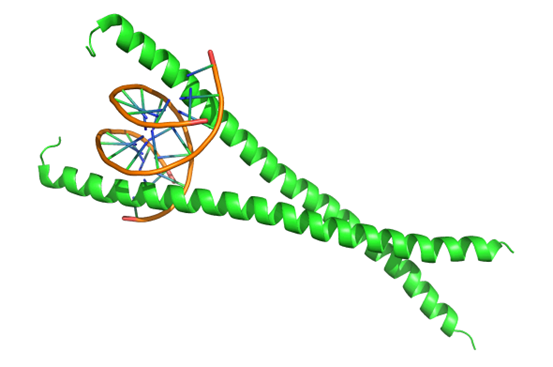
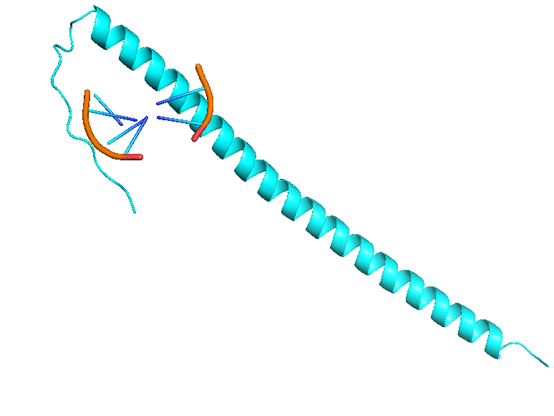
8RZV
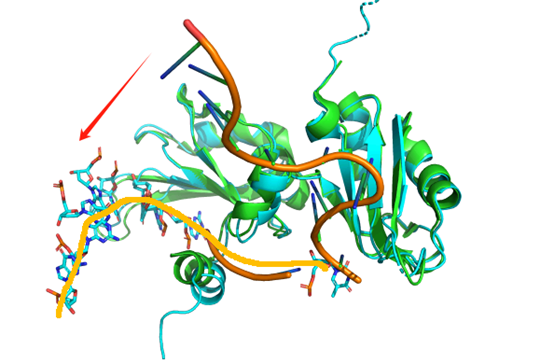
8S1W
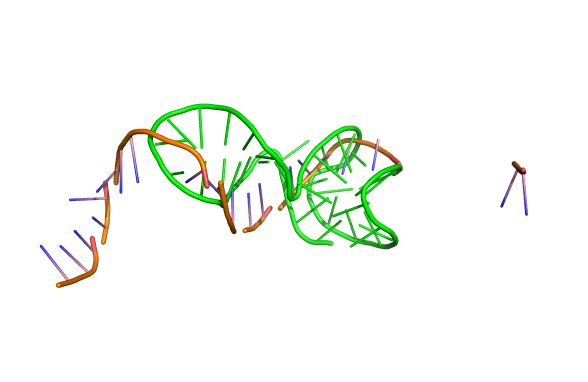
8U0O
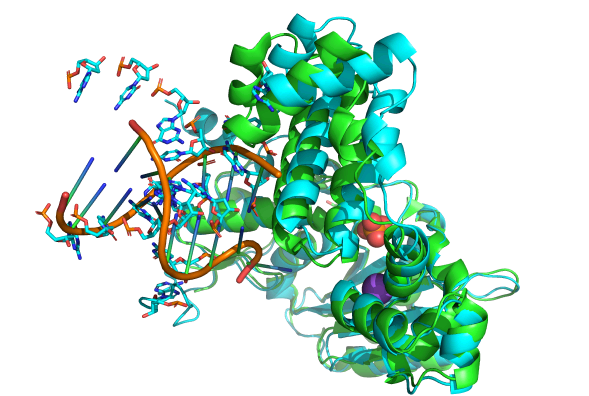
8U8L

8VXA
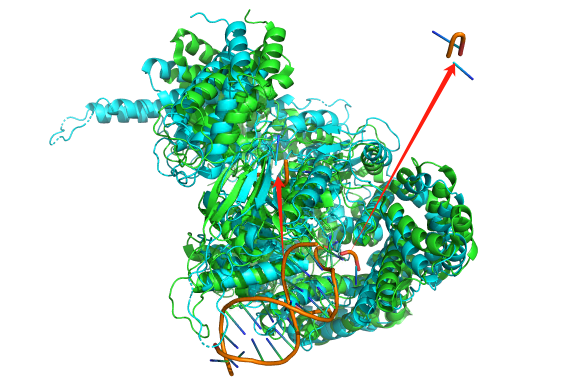
8XA9