二、在北极星集群运行alphafold2.2
在北极星集群运行alphafold2.2:
一、创建并进入lustre3文件夹(lustre1/2/4 gpfs1/2/3,运行比较慢)
mkdir /lustre3/groupname/yourname
二、使用GPU
1、单GPU执行:
A、单体
四卡GPU:alphafold2.2-g4c your.fasta
双卡GPU:alphafold2.2-g2c your.fasta
B:多聚体
四卡GPU:alphafold2.2-g4c your.fasta multimer
双卡GPU:alphafold2.2-g2c your.fasta multimer
--------------------------------------------------------------------
例如: alphafold2.2-g4c 1tce.fasta
daemo:
1) 1TCE daemo:运行命令: alphafold2.2-g4c daemo_1tce
daemo pdb文件 在example/1tce.pdb ![]() 1tce.pdb(点击下载 ,fasta文件 https://www.rcsb.org/fasta/entry/1TCE/display )
1tce.pdb(点击下载 ,fasta文件 https://www.rcsb.org/fasta/entry/1TCE/display )
2) 运行自带的daemo: alphafold-g4c daemo
one GPU maybe better!! lustre3 maybe better!!!!!!!!!!!
2、更多 GPU 使用(不建议使用!!!大序列>1200以上未必能快,详细见后面表格):
双卡:alphafold2.2-g4c 2 1tce.fasta
三卡: alphafold2.2-g4c 3 1tce.fasta
四卡: alphafold2.2-g4c 4 1tce.fasta
双卡服务器双卡: alphafold2.2-g4c 2 1tce.fasta
三、使用CPU:
1、cn_nl 分区
alphafold2.2-cnnl 1tce.fasta
2、cn-short分区
alphafold2.2-cns1tce.fasta
3、多聚体
alphafold2.2-cnnl 1tce.fasta multimer
alphafold2.2-cns 1tce.fasta multimer
注意:cnnl分区使用14个核,cns使用10个核,可以自己更改生成的job.srb+number文件
四、生成的文件:
第一次运行,比如alphafold2.2-g4c daemo_1tce
1、会拷贝生成af2.2_yourname_time的文件夹,如af2.2_005844
2、生成job.srp+时间的文件,多个结构预测建议修改后直接提交(sbatch job.srp+时间)这样可能会快很多很多,第一次生成文件后删掉提交任务就可以直接改用自己修改后的提交脚本
[chenf@login12 test2]$ cat job.srp074306
#!/bin/bash
#SBATCH -J alp005844
#SBATCH -p gpu_4l
#SBATCH -N 1
#SBATCH -o alp005844_%j.out
#SBATCH -e alp005844_%j.err
#SBATCH --no-requeue
#SBATCH -A chenf_g1
#SBATCH --qos=chenfg4c
#SBATCH --gres=gpu:1
#SBATCH --overcommit
#SBATCH --mincpus=7
hosts=`scontrol show hostname $SLURM_JOB_NODELIST` ;echo $hosts
echo CUDA_VISIBLE_DEVICES=$CUDA_VISIBLE_DEVICES
data=/lustre3/alphafold/alphafold2.2/dataset/
#data=/lustre3/alphafold/alphafold2.2/dataset/
#data=/lustre2/alphafold-main/alphafold-2.2.0/dataset
#data=/gpfs1/files_share/alphafold-download/alphafold-2.2.0/dataset
source /appsnew/source/Anaconda3-2022.05-local.sh
source /appsnew/source/cuda-11.0.3.sh
cd /lustre3/chenf/testaf2.2/af2.2_005844
conda activate alphafold2.2
time bash run_alphafold.sh -d $data -o dummy_test/ -f 1tce.fasta -t 2023-02-13 -a $CUDA_VISIBLE_DEVICES
.........
##############
注释一:其中data为所下载的各种数据文件,在哪个文件系统运行就会对应哪个文件系统,比如这次运行在lustre3上就是/lustre3/alphafold/alphafold2.2/dataset/
注释二:run_alphafold.sh可以更改的:
见链接:https://github.com/kalininalab/alphafold_non_docker
注释三:使用GPU比CPU快1倍,1tce的案例单卡GPU运行时间为20??分钟左右(2-4卡也是20分钟),CPU运行时间是37??分钟左右
注释四:输出在err,里面有总速度和每步速度
注释五:多个结构预测的时候,可以复制并修改job.srp074306红色字体,这样第二次以后的计算会快很多很多:
time bash run_alphafold.sh -d $data -o dummy_test/ -f 1tce.fasta -t 2023-02-13 -a $CUDA_VISIBLE_DEVICES
注意改红色的字体,第二次计算的时候会快很多:
改后的文件为job.srp074306:
#!/bin/bash
#SBATCH -J alp005844
#SBATCH -p gpu_4l
#SBATCH -N 1
.........见前面
time bash run_alphafold.sh -d $data -o dummy_test/ -f 1tce.fasta -t 2023-02-13 -a $CUDA_VISIBLE_DEVICES
time bash run_alphafold.sh -d $data -o dummy_test/ -f 6fwt.fasta -t 2023-02-13 -a $CUDA_VISIBLE_DEVICES
time bash run_alphafold.sh -d $data -o dummy_test/ -f 5fwy.fasta -t 2023-02-13 -a $CUDA_VISIBLE_DEVICES
........
########################
然后使用sbatch提交 sbatch job.srp074306
注释六:
Usage: run_alphafold.sh
Required Parameters:
-d
-o
-m
-f
-t
Optional Parameters:
-b
proteins (default: 'False')
-g
-a
-p
3、 /gpfs1/files_share/alphafold/alphafold-ex/alphafold20210728/example/下有pdb结构文件1tce.pdb
4、结果文件夹在af2.2_*/dummy_test 下(*为生成的时间)
features.pkl
ranked_{0,1,2,3,4}.pdb
ranking_debug.json
relaxed_model_{1,2,3,4,5}.pdb
result_model_{1,2,3,4,5}.pkl
timings.json
unrelaxed_model_{1,2,3,4,5}.pdb
msas/
bfd_uniclust_hits.a3m
mgnify_hits.sto
uniref90_hits.sto
参考:https://github.com/deepmind/alphafold
5、可以使用pymol比较1tce.pdb和 ranked_{0,1,2,3,4}.pdb去align
下载链接:http://221.216.6.54:808/sys_bio_lib/software/pymol/
6、在 /lustre3/groupname/yourname/alphafold_yourname_time/alphafold/data/tools/_ 下
比如 /lustre3/chen/alphafold/test/test2/alphafold_chen_074305/alphafold/data/tools/
hhblits.py
hhsearch.py
hmmbuild.py
hmmsearch.py
jackhmmer.py
kalign.py
utils.py
可以修改这几个文件里面的参数。比如jackhmmer.py:
def __init__(self,
*,
binary_path: str,
database_path: str,
n_cpu: int = 8,
n_iter: int = 1,
e_value: float = 0.0001,
z_value: Optional[int] = None,
get_tblout: bool = False,
filter_f1: float = 0.0005,
filter_f2: float = 0.00005,
filter_f3: float = 0.0000005,
incdom_e: Optional[float] = None,
dom_e: Optional[float] = None,
num_streamed_chunks: Optional[int] = None,
streaming_callback: Optional[Callable[[int], None]] = None):
8、daemon_1tce和结果展示:
在lustre3上,用1个GPU卡跑是20分钟,用默认CPU跑是37分钟
在lustre3你的文件夹下运行:
alphafold-g4c daemo_1tce
结果见上面描述:
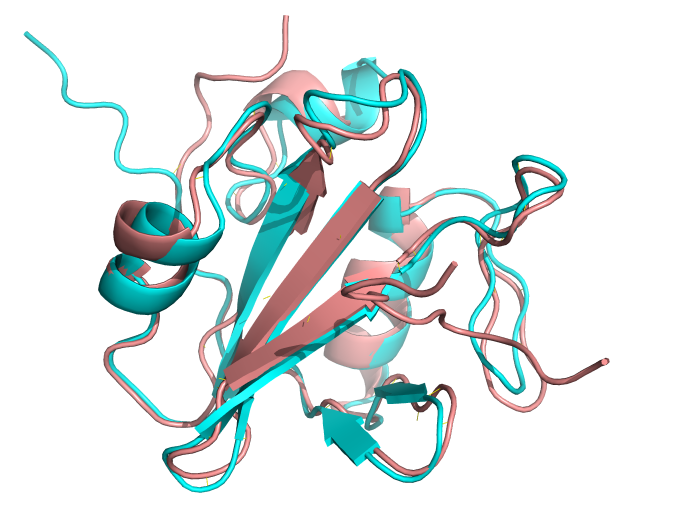
浅棕色是1TCE的晶体结构,青色是预测的结构。在lustre3上,用1个GPU卡跑是20分钟,用默认CPU跑是37分钟
9,不能超过一个蛋白链:
如:
>2JO9_1|Chain A|Itchy E3 ubiquitin protein ligase|Mus musculus (10090) GAMGPLPPGWEKRTDSNGRVYFVNHNTRITQWEDPRS >2JO9_2|Chain B|Latent membrane protein 2|null EEPPPPYED
More than one input sequence found in /lustre3/chen/alphafold/test/test2/alphafold_chen_005/2JOV.fasta