-
三、在北极星集群运行alphafold2.3-运行
在北极星集群运行alphafold2.3:一、创建并进入lustre3文件夹(lustre1/2/4 gpfs1/2/3,运行比较慢)mkdir /lustre3/groupname/yourname二、使用GPU1、单GPU执行(自动识别多聚体):四卡GPU:af2.3-g4c your.fasta 双卡GPU:af2.3-g2c your.fasta -----------------
2023-06-03 admin 61
-
一·、在北极星集群上提交RoseTTAFold-All-Atom------PDB格式
在北极星集群上提交RoseTTAFold-All-Atom一、命令格式RFAA.run [–pymol] [pdb|fasta] [small.pdb|small.mol2]格式说明1、 直接跑 pdb结构 : RFAA.run 1tce.pdb 如果想只跑A链 RFAA.run 1tce.pdb:A 2、将所有的蛋白、DNA、RNA合成一个fasta文件 : RFAA.run in
2024-04-13 admin 0
-
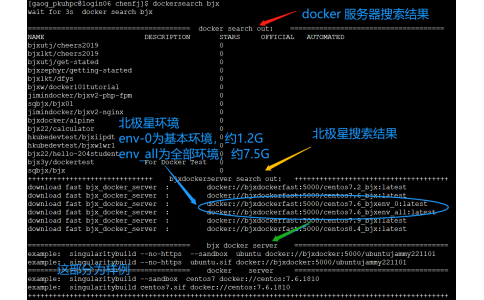
二、singularity使用北极星环境的镜像--运行北极星程序-matlab-R-mpi编译器
二、使用北极星环境的镜像--运行北极星程序-matlab-R-mpi编译器北极星的运行的环境为centos:7.6.18101、搜索镜像;推荐在login06下载为内网:运行:dockersearch bjx # 或者dockersearch centos运行结构如下图:其中login06:docker://bjxdockerfast:5000/centos7.6_bjxenv_0:latest
2022-12-02 admin 252
-
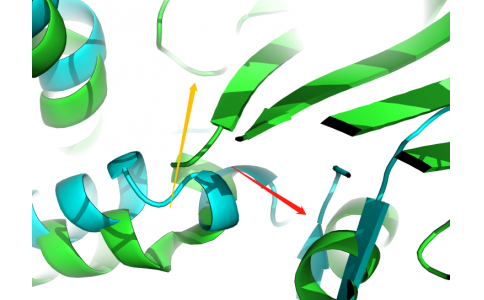
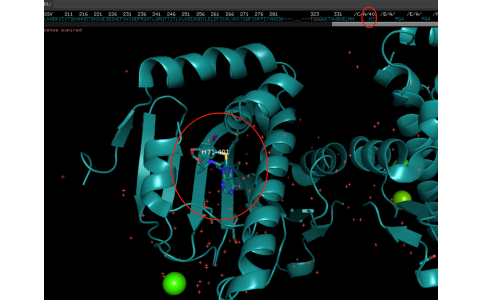
alphafold3蛋白和小分子相互作用(一)8ssv json模板,小分子结构库里有的(2024年11月结构)
使用https://www.rcsb.org/ligand/H71 的smiles格式计算1、获取小分子信息2、打开网页,3、打开https://www.rcsb.org/fasta/entry/8SSV/display fasta文件4、汇总信息、生成json文件,其中ZN和GOL等基础分子(离子),为默认的ccd格式,是有的,不需要提供smiles格式8ssv.json:{ "
2024-11-22 admin 0
-
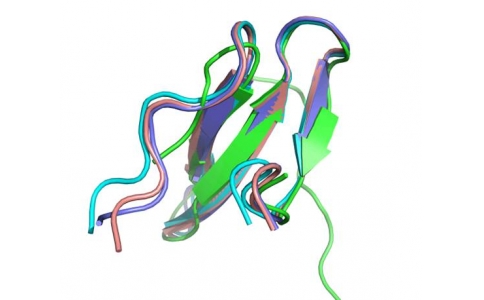
alphafold3蛋白和小分子相互作用(二)1w83 json模板 小分子转换为smiles格式
1、截取1w83的小分子存为pdb文件使用 obabel 转化obabel -ipdb 1w83small.pdb -osmi -O 1w83small.smi 如果是mol2格式或者sdf格式obabel -imol2 1w83small.mol2 -osmi -O 1w83small.smiobabel -isdf 1w83small.sdf -osmi -O 1w8
2024-11-22 admin 0
-
四、在af中使用pymol(一)conda下的pymol安装和创建桌面图标
(一)conda下的pymol安装1、linux下的安装配置环境source /appsnew/source/Anaconda3-2022.05-local.sh创建虚拟环境conda create -n pymol2.5安装pymol2.5版本conda install -c schrodinger -c conda-forge pymol或安装pymol2.4等版本conda install
2023-05-05 admin 34
-
三、在图形界面创建桌面快捷方式-(二)自定义桌面图标启动的matlab版本
进入桌面文件夹:[chen@login25 桌面]# vi Matlab.desktop 输入后面cat命令后的内容[chen@login25 桌面]# cat Matlab.desktop[Desktop Entry]Type=ApplicationName=MatlabGenericName=Matlab 2022bComment=Matlab:The Language of Technic
2023-04-26 admin 53
-
alphafold3蛋白和小分子相互作用(三)8R2K json模板 只有cif格式的结构(2024年11月的结构)
1、查看获取小分子信息直接打开 https://www.rcsb.org/ligand/XPI 下载sdf格式wget https://files.rcsb.org/ligands/download/XPI_ideal.sdfobabel -isdf XPI_ideal.sdf -osmi -O 8r2k.smi[chenfj@login26 testnew01]$ cat 8r2k.
2024-11-22 admin 0