-
alphafold3蛋白和小分子相互作用(二)1w83 json模板 小分子转换为smiles格式
1、截取1w83的小分子存为pdb文件使用 obabel 转化obabel -ipdb 1w83small.pdb -osmi -O 1w83small.smi 如果是mol2格式或者sdf格式obabel -imol2 1w83small.mol2 -osmi -O 1w83small.smiobabel -isdf 1w83small.sdf -osmi -O 1w8
2024-11-22 admin 0
-
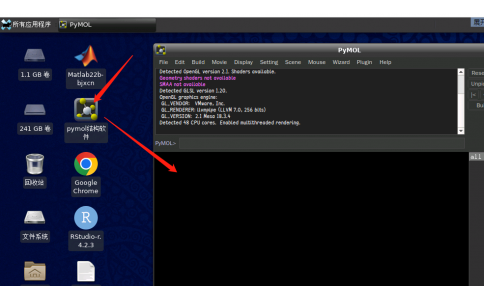
四、在af中使用pymol(一)conda下的pymol安装和创建桌面图标
(一)conda下的pymol安装1、linux下的安装配置环境source /appsnew/source/Anaconda3-2022.05-local.sh创建虚拟环境conda create -n pymol2.5安装pymol2.5版本conda install -c schrodinger -c conda-forge pymol或安装pymol2.4等版本conda install
2023-05-05 admin 34
-
三、在图形界面创建桌面快捷方式-(二)自定义桌面图标启动的matlab版本
进入桌面文件夹:[chen@login25 桌面]# vi Matlab.desktop 输入后面cat命令后的内容[chen@login25 桌面]# cat Matlab.desktop[Desktop Entry]Type=ApplicationName=MatlabGenericName=Matlab 2022bComment=Matlab:The Language of Technic
2023-04-26 admin 53
-
alphafold3蛋白和小分子相互作用(三)8R2K json模板 只有cif格式的结构(2024年11月的结构)
1、查看获取小分子信息直接打开 https://www.rcsb.org/ligand/XPI 下载sdf格式wget https://files.rcsb.org/ligands/download/XPI_ideal.sdfobabel -isdf XPI_ideal.sdf -osmi -O 8r2k.smi[chenfj@login26 testnew01]$ cat 8r2k.
2024-11-22 admin 0
-
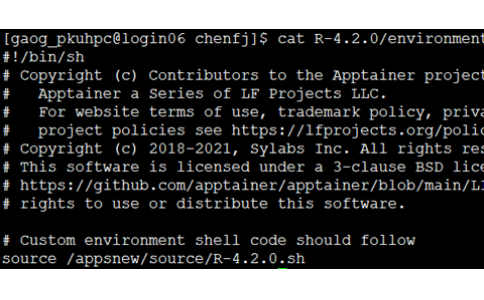
三、查询tag,和使用原生centos挂载使用北极星集群安装软件--singularity修改、打包和运行镜像
三、修改镜像并打包,将/appnews挂载到容器里,这里我们以运行/appsnew/usr/R/R-4.2.0/lib64/R为例这种就可以把集群已经安装好的软件,打包到里面去,然后安转设置别的软件,比如intel编译器等,后面mpi、GPU会讲到1、拉取推荐使用北极星集群dockerserver执行:dockersearch centos输入提示如箭头在login06(比较快):singular
2022-12-05 admin 151
-
alphafold3蛋白和小分子相互作用(四)8IT4 蛋白质和小分子共价json模板
8IT41、获取小分子SKU的cif文件https://www.rcsb.org/ligand/SKU如何查看ccdCodes:下图显示是SKU,连接原子数是29。连接的残基是A链的92位置氨基酸下载 https://files.rcsb.org/ligands/download/SKU.cif2、预先准备好json记住:a、bond必须使用cif不能用smiles,而且得自己合进json文件b、
2024-11-25 admin 0
-
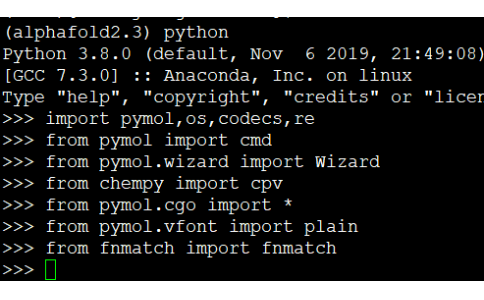
四、在af中使用pymol(二)使用python下载结构、创建和查看pse文件和渲染图片
pse文件为pymol的会话文件;pse 文件类似于PS中的psd文件,方便修改调整。1、进入虚拟环境--alphafold2.3,执行python[chen@login29 etc]# source /appsnew/source/Anaconda3-2022.05-local.sh(base)[chen@login29 etc]$conda activate alphafold2.3(alp
2023-05-06 admin 19
-
四、公共app桌面的图标建立-(一)雷鸟邮件系统
一、需要root权限去完成,如果有公共的app做好后联系管理员后,复制到[root@login26 applications]# pwd/usr/share/applications这个文件二、查找icons图标文件,使用rpm -ql 去查看包已经安装文件[root@login26 applications]# rpm -ql thunderbird |grep -i icons/usr/l
2023-04-26 admin 56